La Plateforme INteractions moléculairesTimone (PINT) offre une expertise unique dans l’analyse thermodynamique et cinétique des interactions moléculaires.
PINT est équipée de microcalorimètres (ITC et DSC), de nanoDSF, de spectrophotomètres et d’une Ultracentrifugeuse Analytique (UCA), et d’un biocapteur optique basé sur la résonance plasmonique de surface (SPR).
Ces techniques ont toutes pour objet de caractériser les interactions moléculaires (protéines, acides nucléiques, sucres, lipides, petites molécules, peptides, protéoliposomes, particules virales, etc…). Elles ont en commun l’absence d’utilisation de réactifs marqués, la simplicité et la rapidité de mise en œuvre. La complémentarité de ces techniques est largement référencée pour la caractérisation des interactions protéines-ligands
Cette plateforme est ouverte à la communauté de chercheurs académiques comme industriels.
La plateforme PINT est localisée au 3eme étage de l’aile verte de la faculté de médecine.
Pôle Microcalorimétrie
Responsable : Philipp TSVETKOV, François DEVRED
Equipements à disposition : 3 microcalorimètres: ITC200, VP-ITC et VP-DSC permettant à la fois de mener des études de calorimétrie de titration isotherme (ITC) et de calorimétrie à balayage de température (DSC), ainsi que d'un nanoDSF NT.Plex.
L'ITC permet de caractériser les interactions intermoléculaires en obtenant leur profil thermodynamique complet : constante d'affinité, stœchiométrie, enthalpie et entropie.
La DSC et le nanoDSF permettent de déterminer la thermostabilité (5-120 °C) de protéines, ADN, micelles ... ce qui donne notamment des indications sur la structure tertiaire des molécules et les interactions moléculaires. Le nanoDSF par l'utilisation de capillaires peut aussi être utilisé pour faire du screening d'interactions.
Parallèlement à notre activité de prestation de service, nous menons une activité R/D ayant pour objectif la mise au point d'une nouvelle méthode diagnostique par profilage calorimétrique des biofluides. En collaboration avec l'équipe 8 de l'INP et le Service de Neuro-oncologie de La Timone (AP-HM) nous travaillons à la mise au point d'une méthode de suivi des glioblastomes (Tsvetkov et al Oncotarget 2018). En partenariat avec l'équipe QARMA du Laboratoire Informatique et Systemes, AMU et la société EURANOVA toutes deux spécalistes de l'apprentissage automatique (machine learning), nous developpons une approche universelle visant à généraliser cette méthode à d'autres maladies du système nerveux (Alzheimer, Parkinson, multiple sclerosis …).
Pôle Ultracentrifugation Analytique
Responsable : Pascale BARBIER
Equipement à disposition : Ultracentrifugeuse Analytique Beckman XLA
L'UltraCentrifugation Analytique (UCA) permet l’étude du comportement des macromolécules natives et en solution, soumises à une force centrifuge. A l’aide d’un système de détection qui permet de mesurer la concentration en macromolécule en fonction du rayon de centrifugation, nous pouvons effectuer deux types d’expériences qui vont conduire à l’obtention d’informations complémentaires :
La vitesse de sédimentation permet d’obtenir le coefficient de sédimentation, la forme, l’état associatif (monomère, dimère, trimère,…) d’une macromolécule. Elle peut aussi mettre en évidence la formation d’un complexe et permettre la détermination de sa constante d’affinité.
L’équilibre de sédimentation permet d’obtenir la masse, l’état associatif (monomère, dimère, trimère,…) et de mettre en évidence la formation d’un complexe macromoléculaire.
Pôle Résonance Plasmonique de Surface (SPR)
Responsable : Géraldine FERRACCI
Equipements à disposition : Biacore T200, Biacore 3000 et ProteOn XPR36
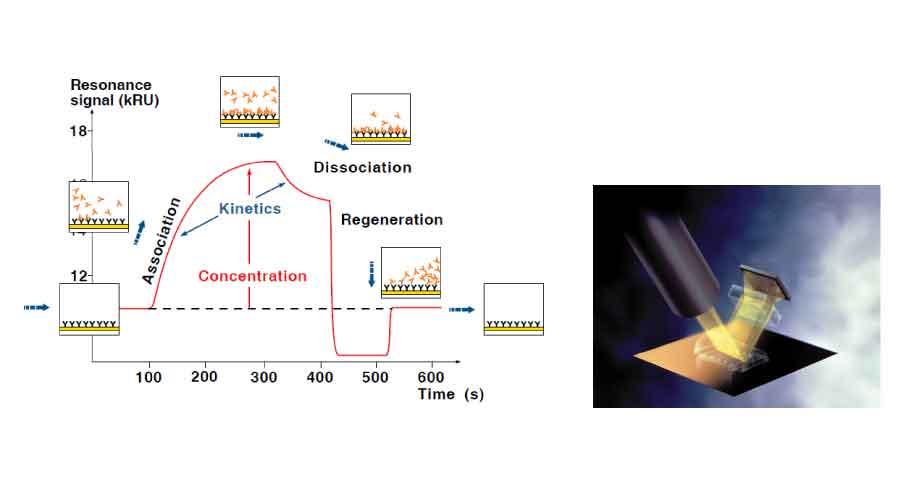
Les systèmes SPR sont conçus pour visualiser en temps réel les interactions entre biomolécules non marquées. Un des réactifs, le ligand, est retenu sur la sensor chip et les autres partenaires de l’interaction, appelés analytes, sont injectés à un débit constant à l'aide d'un circuit microfluidique. Le principe de la SPR permet de suivre à tout moment les changements de masse à la surface de la sensor chip, induits par la formation puis la dissociation des complexes moléculaires.
L’exploitation de la mesure en temps réel va permettre d’obtenir les constantes cinétiques d’association (ka) et de dissociation (kd) et d’en déduire l’affinité (KD) de l’interaction. Avec un mode d’analyses automatisées, parallélisées et miniaturisées, les biocapteurs optiques SPR permettent en outre de définir les constantes thermodynamiques, la concentration active d’un analyte, des cartographies épitopiques ou d’étudier des molécules en milieux complexes et de réaliser de la micropurification et récupération de partenaires inconnus.
Pole CAPM analyse protéomique
Responsable : Maya Belghazi, maya.belghazi@univ-amu.fr
Equipement à disposition : Micro-HPLC Smart System BioRad ; Ultraflex II TOF-TOF Bruker ; Q-TOF Ultima Waters ; TECAN EVO 100 ; Nano UHPLC DIONEX 3000.
La spectrométrie de masse est une technique physique d’analyse qui permet de détecter et d’identifier des molécules par mesure de leur masse et de caractériser leur composition chimique. Services proposés : Masse entière ; Identification PMF ou MSMS ; Quantification iTRAQ ; Séquençage de novo.
Partenaires:
Voici une liste non exhaustive des équipes ayant fait appel à la plateforme PINT :
Marseille medical genetics, Inserm UMR U1251, Marseille (Marc Bartoli) - Institut de Chimie de Nice, UMR CNRS 7272, Nice (Rachid Benhida) - Centre européen de recherche en imagerie médicale, Marseille (Benjamin Guillet) - INP, Institut de NeuroPhysiopathologie, UMR CNRS 7051, Marseille (Francois Devred, Emeline Tabouret) - CRCM, Centre de recherche en cancérologie de Marseille, Inserm U1068, Marseille (Joseph Ciccolini, Eddy Pasquier) - Laboratoire d'Ingénierie des Systèmes Macromoléculaires,UMR CNRS 7255, Marseille (James Sturgis) - Grenoble Institut Neurosciences, Inserm UMR U1216, Grenoble (Isabelle Arnal) - Architecture et Fonction des Macromolécules Biologiques, CNRS UMR 7257, Marseille, (Sonia Longhi) - Institut de Microbiologie de la Méditerranée, Laboratoire de chimie Bactérienne, CNRS UMR 7283, Marseille, (Romé Voulhoux) - Institut de Microbiologie de la Méditerranée, Bioénergétique et Ingénierie des Protéines, CNRS UMR 7281, Marseille, (Brigitte Gontéro)

