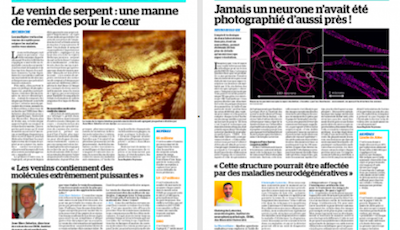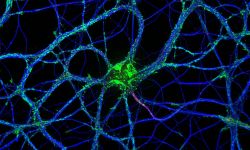

L'équipe NeutoCyto: “fonctions et dysfunctions du cytosquelette neuronal” a démarré en 2017 dans le cadre du programme ATIP du CNRS. Nous sommes des biologistes cellulaires du neurone: nous voulons comprendre comment les neurones sont organisés au niveau cellulaire. Comment se différencient-ils, puis construisent et maintiennent leur arborisation complexe? Comment établissent-ils et conservent-ils leur polarité, avec l'axone et les dendrites permettant d'envoyer et de recevoir des signaux? De nombreux processus contribuent à cette organisation: élaboration de l'architecture cellulaire (grâce au cytosquelette), transport de protéines à l'intérieur de la cellule (avec protéines de diffusion et motrices), ségrégation dans des compartiments distincts (tels que l'axone, les synapses, les épines dendritiques…). Nous appliquons des techniques de microscopie avancées pour observer directement les assemblages moléculaires à l'échelle nanométrique dans les neurones, révélant ainsi comment ils organisent le neurone et façonnent sa physiologie.
« NeuroCyto: fonctions et dysfonctions du cytosquelette neuronal » est une équipe ATIP au sein de l’Institut de NeuroPhysiopathologie (INP, CNRS-Aix Marseille Université UMR 7051). L’équipe comporte actuellement six personnes, et nous accueillons des étudiants tout au long de l’année. N’hésitez pas à nous contacter si vous êtes intéressé! Notre but est de composer une équipe dynamique pour faire la meilleure science possible en cultivant l’ouverture, l’échange, et l’enthousiasme pour les découvertes petites ou grandes. Nous nous intéressons à la façon dont les neurones s’organisent en tant que cellules : comment font-ils pour se différencier, puis développer et maintenir leur arborisation complexe ? Comment maintiennent-ils leur polarité, avec un axone et des dendrites qui permettent d’envoyer et de recevoir l’information ? La construction de l’architecture cellulaire (grâce au cytosquelette), les mécanismes du transport des protéines (moteurs, diffusion), la compartimentation en éléments distincts (axone, synapses, épines dendritiques…) : de nombreux processus encore largement mystérieux contribuent à cette organisation. Pour mieux les comprendre, l’équipe NeuroCyto applique de nouvelles méthodes de microscopie, notamment la microscopie de super-résolution qui permet d’aller observer les assemblages moléculaires à l’échelle nanométrique directement dans les cellules pour mieux comprendre le fonctionnement neuronal.
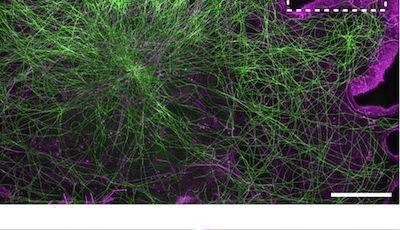
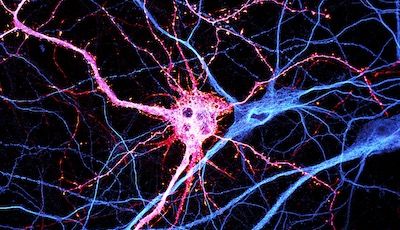
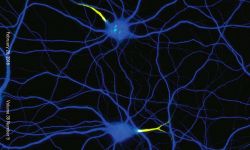
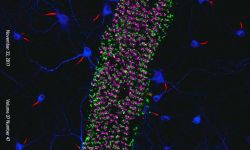
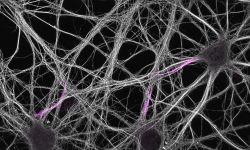
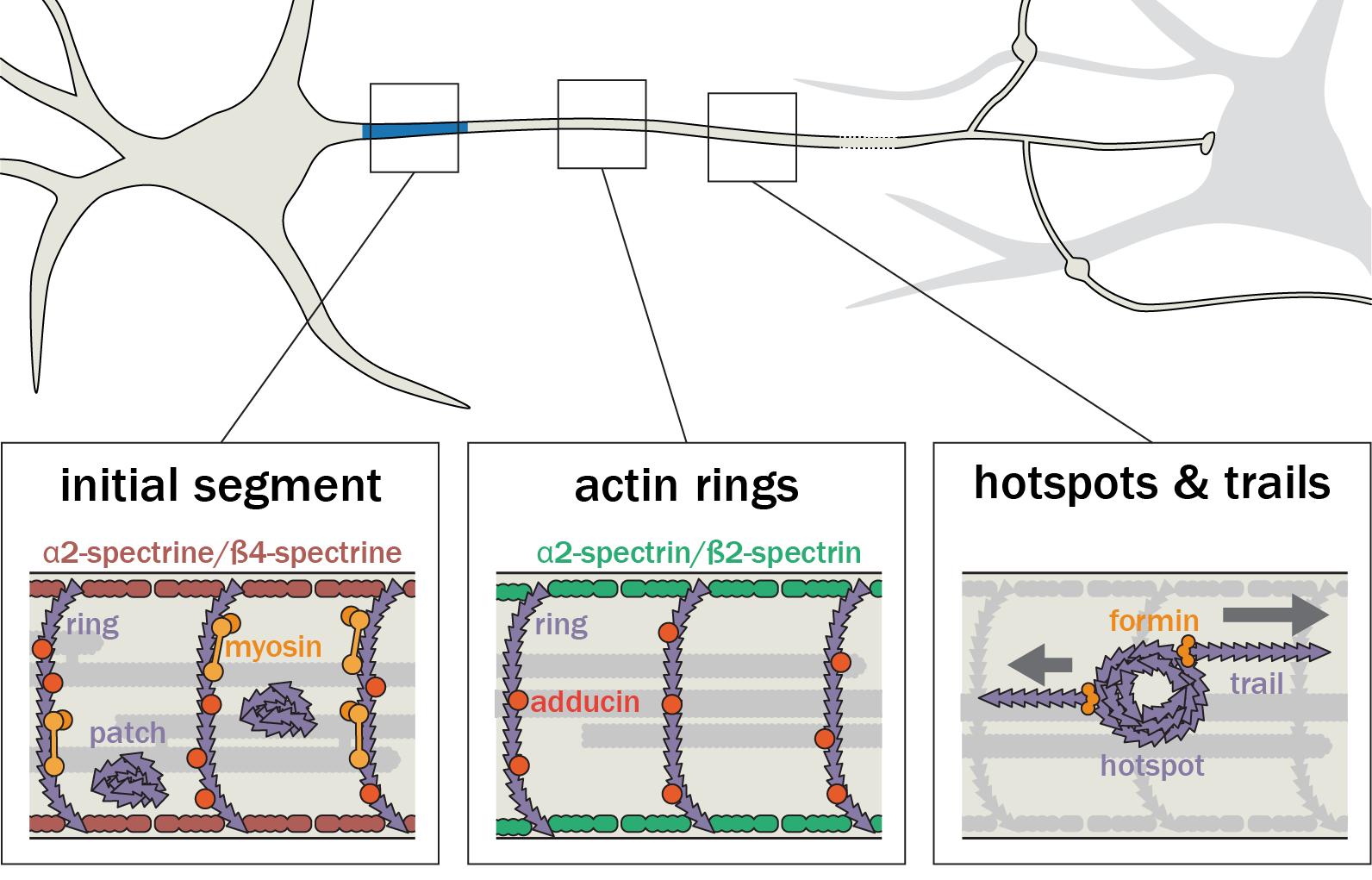
Nous nous concentrons actuellement sur l'organisation de l'actine au sein des axones. Nous avons découvert, avec d'autres structures, plusieurs nouvelles structures d'actine axonale, notamment des anneaux d'actine sous-membranaires et les hotspots et trails d'actine intra-axonale. Nous voulons comprendre les fonctions de ces structures et leur pertinence pour des processus physiologiques tels que le transport axonal et le bon fonctionnement des boutons présynaptiques. Pour ce faire, nous tirons parti du modèle de neurones d'hippocampe de rat en culture à faible densité afin de pouvoir visualiser la morphologie complexe des cellules et de suivre des axones individuels. Nous combinons imagerie de cellules vivantes, manipulations ciblées et microscopie à super-résolution pour relier le comportement dynamique des composants axonaux à l'organisation nanoscopique du cytosquelette.
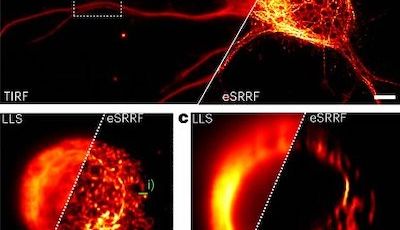
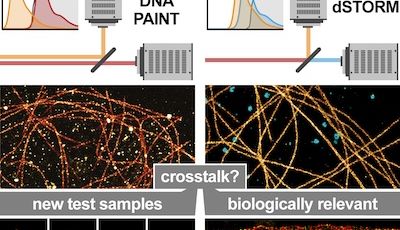
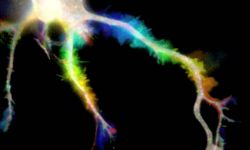
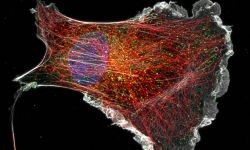
Nous consacrons d'importants efforts à la mise en œuvre de stratégies innovantes pour l'étiquetage, l'observation et l'analyse de ces processus. Nous sommes des spécialistes de la microscopie de localisation de molécule unique (SMLM) et nous continuons à faire progresser les méthodes, en mettant en œuvre et en testant de nouvelles techniques dès leur apparition. Cela inclut des méthodes de marquage pour une acquisition hautement multiplexée telles que DNA-PAINT, des stratégies d’acquisition automatisées et de nouveaux algorithmes d’analyse d’images.
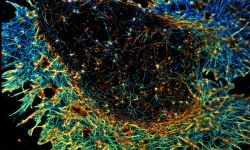
Enfin, nous explorons la pertinence physiopathologique de notre découverte en étudiant des modèles cellulaires de la maladie d’Alzheimer, dans la mesure où le transport axonal et la fonction présynaptique sont compromis aux premiers stades de la maladie. En utilisant nos connaissances en biologie cellulaire, nous souhaitons mieux comprendre pourquoi cela se produit en étudiant l'organisation à l'échelle nanométrique de l'axone dans les modèles de maladie d'Alzheimer: modèles marins, mais aussi neurones humains obtenus à partir de cellules souches pluripotentes induites.